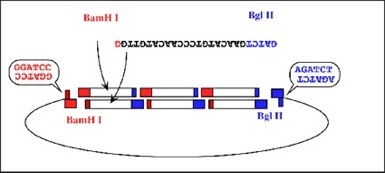
[原理]
合成するOligonucleotideの末端にBgl IIとBamH Iなど、突出末端部の配列が同じ酵素サイトを付加し、ライゲーションさせることによって(図参照)コピー同士が切り離せないフラグメントを作製することができる。
[方法]
★ Oligonucleotideの場合、plasmidを扱うときよりも標的サイトが多いので、各修飾、制限酵素反応は十分な酵素量を加える必要がある。
1. Oligonucleotideの合成
図のように両末端に酵素サイトの一部をつけたセンス鎖とアンチセンス鎖の2種類を合成する
2. Oligonucleotideのアニーリング
↓下記に示すように反応液を調整する
sense oligonucleotide 5 ul (5 ug)
antisense oligonucleotide 5 ul (5 ug)
(Total 10 ul)
↓ Denature 100℃ 30 sec.
↓ ゆっくり室温まで戻す(30 min.)
↓ 12% PAGEでアニーリングしているかcheck
3. 末端のリン酸化
↓下記に示す反応液を調整する
(2)のsample 10 ul
T4 PNK *1 1 ul
ATP (2.5 mM) 8 ul
10×Protruding End Kinase Buffer 2 ul
(Total 21 ul)
↓ 37℃ 2 hr以上
↓ 65℃ 5 min.
4. フラグメントのLigation
↓下記に示す反応液を調整する *2
(3)のsample 21 ul
T4 DNA Ligase *3 2 ul
2.5 mM ATP 18 ul
10×Ligation Buffer 2 ul
(Total 41 ul)
↓ 16℃ O/N
↓ 65℃ 5 min.
↓ on ice 3 min.
↓ Fill up to 200 ul (add 160 ul of TE)
↓ フェノールクロロホルム処理
↓ エタノール沈殿
↓ TE 44 ul に溶かす
5. 制限酵素カット *4
↓下記に示す反応液を調整する
(4)のsample 44 ul
BamH I 7 ul
Bgl II 3 ul
10×High Buffer 6 ul
(Total 60 ul)
↓ 37℃ 2 hr
↓ 12% PAGEでラダーになっているかcheck
6. フラグメントの切り出し(ゲルからのDNA切り出しprotocol 参照)
↓ 12% PAGEで泳動し、各高さのバンドを切り出す
↓ フェノールクロロホルム処理、エタノール沈殿後、15 ulのTEに溶かす
7. BamH I / Bgl II vectorにサブクローニング(サブクローニングprotcol参照)
8. プラスミド精製(プラスミド精製protcol参照)
9. BamH I / Bgl IIで制限酵素カットcheck
10. シークエンスにてcheck
*1) T4 Polynucleotide Kinase (PNK-111/TOYOBO)
*2) リン酸化で用いたBufferをそのまま使えるので、半量のBufferを継ぎ足すだけでよい。
*3) T4 DNA Ligase (LGA-111/TOYOBO)
*4) フラグメント同士がBamH I/BamH IやBgl II/Bgl IIで逆向きにLigationしているのを切断する。